Atelier ChIP-seq
Contexte
Cet atelier a lieu dans le cadre de
l'Ecole de bioinformatique AVIESAN 2014, Station Biologique, Roscoff .
Flowchart général des étapes
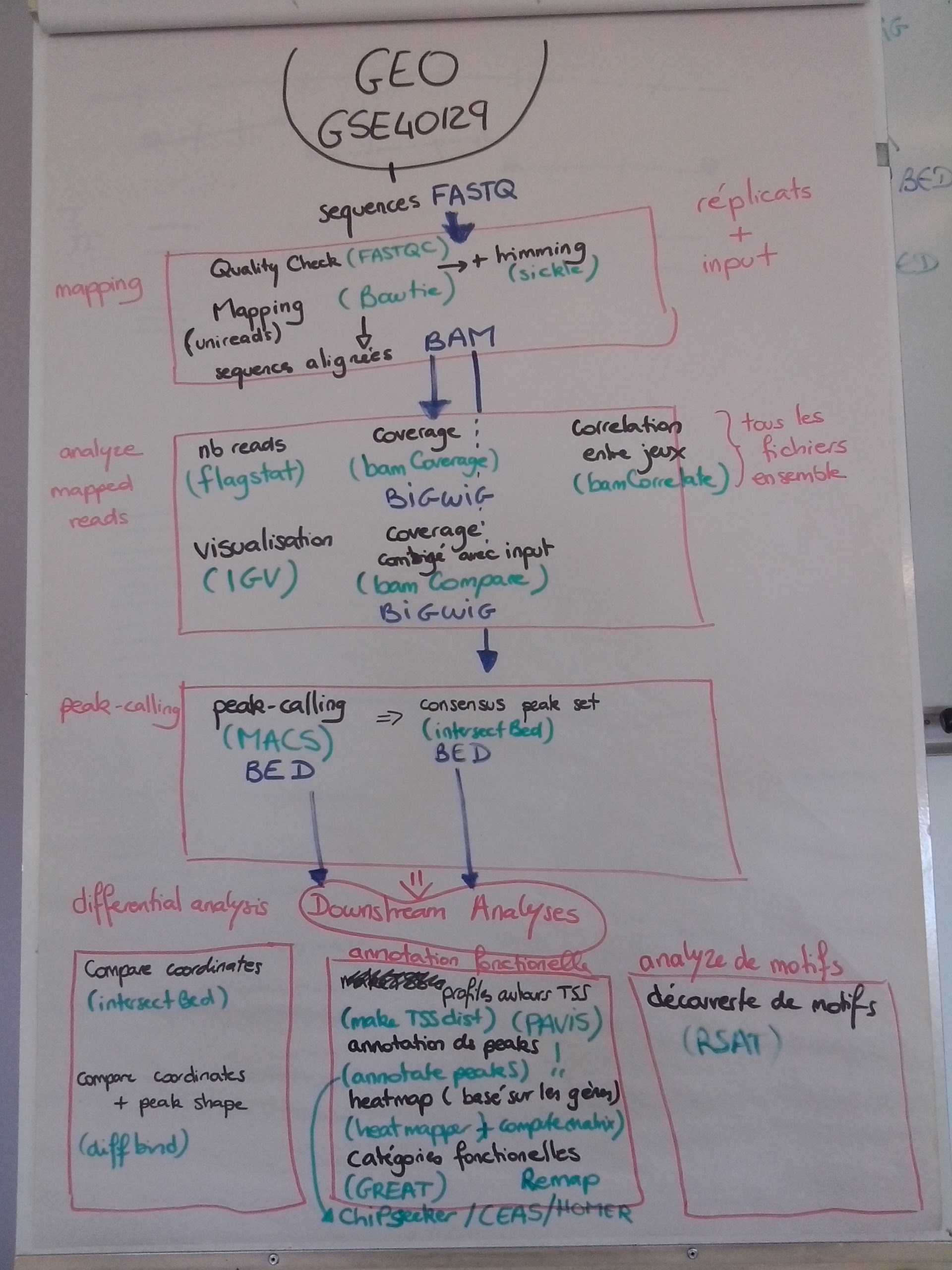
Organisation
| Session |
Date |
De |
A |
Titre |
Aspects théoriques (diapos) |
Travaux pratiques |
| 1 |
Mardi 07/10 |
14:00 |
17:30 |
ChIP-seq : normalisation et peak-calling |
- Qualité et normalization des données de ChIP-seq
- Introduction sur le peak-calling
- Diversité des approches
- les outils MACS et SICER
- Analyse différentielle
|
- Alignement des reads, quality check Etapes préalablement suivies pour obtenir les fichiers BAM (reads alignés). Pour des raisons de temps, nous ne pouvons pas faire des étapes lors de l'atelier. Nous encourageons les participants à suivre ce tutorial de manière autonome.
- Peak Calling
|
| 2 |
Mercredi 08/10 |
8:30 |
10:00 |
Annotation, visualisation et intégration de données ChIP-seq |
- Annotation génomique (gènes les plus proches, profil autour du TSS)
- Annotation fonctionnelle (enrichissement en termes fonctionnels, GREAT)
- Intégration de données (lien ChIP-seq / RNA-seq, MedIP-seq, …)
|
- Tutorial [PDF]
- Visualisation de l’enrichissement
- Gènes associés aux peaks
- Enrichissement en termes fonctionnels (GREAT)
|
| 3 |
Mercredi 08/10 |
10:30 |
12:30 |
Détection de motifs dans les pics |
- Découverte de motifs
- Comparing discovered motifs with transcription factor databases.
- Predicting binding sites
|
- Analyse de motifs dans les pics de ChIP-seq
|
Formateurs
- Carl Herrmann1
- Matthieu Defrance2
- Denis Puthier3
- Morgane Thomas-Chollier4
- Cancer Regulatory Genomics group, Division of Theoretical Bioinformatics, DKFZ and IPMB, Universität Heidelberg,
Web: website
Build. TP3 - DKFZ Heidelberg (B080) Im Neuenheimer Feld 580 D-69120 Heidelberg.
- Laboratory of cancer epigenetics
Université; Libre de Bruxelles
Faculté de Médecine CP614. Route de Lennik, 808. 1070 Bruxelles, Belgium
- Lab. Technological Advances for Genomics and Clinics (TAGC)
Web: website
Aix-Marseille Université (AMU).
INSERM Unit U1090, 163, Avenue de Luminy, 13288 MARSEILLE cedex 09. France
-
CSB group, Institut de Biologie de l'École Normale Supérieure
Institut de Biologie de l'École Normale Supérieure, 46 rue d'Ulm F-75230 Paris cedex 05 . France
Web: website

